La découverte et la mise sur le marché d'un nouveau médicament reste encore un processus long et risqué : 10 à 15 ans en moyenne, avec taux d'échec important et un coût moyen de 2 milliards d'euros. « Lors de la phase de découverte, il y a des étapes exploratoires pour comprendre la pathologie que l'on cherche à traiter et identifier une cible thérapeutique, puis une phase de criblage de différentes molécules en vue de moduler l'activité de la cible. Ensuite seulement, on entre en phase de tests pré-cliniques, puis cliniques », explique Jeremy Grignard, chercheur et data scientist à l'Institut de Recherches Servier. L'un des enjeux pour les laboratoires pharmaceutiques est donc d'augmenter le taux de succès des candidats médicaments qui progresseront dans toutes les phases cliniques chez l'humain.
C'est sur ce sujet que le laboratoire Servier, spécialisé dans les maladies cardiovasculaires, les cancers, le diabète, les maladies immuno-inflammatoires et neuro-psychiatriques, a accueilli Jeremy Grignard pour une thèse Cifre en 2017, en partenariat avec Inria-Saclay. Dans la continuité de ses travaux, l'Institut de recherche du groupe a monté en 2022 un département de science des données, notamment pour explorer le potentiel des technologies d'apprentissage automatique et profond.
Intégrer dans un même modèle des concepts très hétérogènes
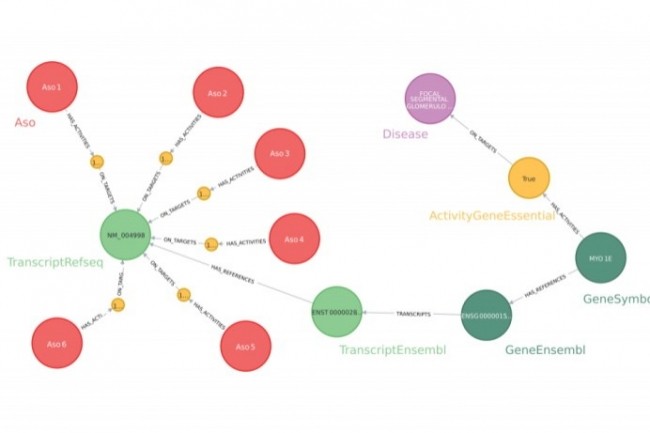
Au total, les chercheurs pharmaceutiques travaillent avec près d'une cinquantaine de sources de données différentes lors des phases de découvertes. « Hormis cette volumétrie très conséquente, les concepts pharmaco-biologiques sont très hétérogènes. Le projet Pegasus avait pour objectif d'intégrer et de fédérer l'ensemble de ces données dans un graphe de connaissances. Il inclut un peu plus de 46 millions d'entités de 66 étiquettes différentes et plus de 330 millions de relations, de 14 types différents », témoigne le data scientist. Pour construire des graphes, celui-ci a choisi de travailler avec Neo4J, à la fois pour la flexibilité du modèle de données, qui répondait aux enjeux d'évolutivité dans le temps, mais aussi pour ses capacités de stockage et de requêtage efficaces, ainsi qu'une préparation de données facilitée. « Pegasus a été conçu pour donner des réponses très rapidement, afin d'aider les chercheurs à tester et confronter différentes hypothèses » explique Jeremy Grignard. « Répondre à une question scientifique donnée revient à traverser un ensemble de chemins dans le graphe de connaissances. »
Pegasus est alimenté par des données brutes, qui proviennent de bases de données scientifiques ou sont issues de modèles d'intelligence artificielle. Ensuite, les utilisateurs y accèdent à travers des applications et des rapports d'analyse automatisés. Parmi les premiers cas d'usage explorés figure la recherche d'oligonucléotides antisens (ASOs), qui permettent de moduler une protéine cible. « Sur 100 000 ASOs conçus pour une cible thérapeutique, nous en avons priorisé 784 que nous avons validés expérimentalement, en vérifiant par exemple ceux qui risquaient d'avoir des activités sur d'autres cibles que celle définie, à écarter en raison des risques d'effets secondaires », illustre Jeremy Grignard.
Un taux de hit de 15%
Historiquement, le criblage de molécules chimiques était réalisé de façon aléatoire parmi des millions de candidates. Sur quelques expériences, la solution basée sur les graphes de connaissances a été comparée avec les résultats obtenus par le processus classique, pour observer si elle permettait d'enrichir le nombre de hits. « Nous avons identifié 984 molécules avec un taux de hit à 15%, alors que par défaut il tourne autour d'1% », indique Jeremy Grignard. La solution est désormais utilisée dans le cadre de plusieurs projets thérapeutiques, afin d'augmenter le taux de succès et la qualité des molécules sélectionnées.
Commentaire